Número actual.
Enero-Marzo 2026 vol. 77-1
Portada
Desde el Comité Editorial
Embarazo adolescente, una visión integral
El embarazo adolescente es un asunto de la familia, la escuela y el Estado
El papel de la dinámica familiar en el embarazo adolescente
La búsqueda de afecto y familia en el contexto del embarazo adolescente
Transformación de la identidad en el embarazo adolescente
Hormonas, metabolismo y bioquímica durante el embarazo de una adolescente
Las barreras que dificultan una buena lactancia en madres adolescentes
La importancia de la salud bucodental durante el embarazo de las adolescentes
Alimentación y hábitos saludables en las adolescentes embarazadas
La programación de la vida desde el vientre: nutrición, genética y epigenética
Mitos comunes sobre la anticoncepción tras el embarazo adolescente
Microorganismos que ayudan a descontaminar agua con arsénico
Marcadores moleculares del ADN: huellas genéticas que revelan secretos de los seres vivos
Noticias de la Academia Mexicana de Ciencias
La degradación ambiental de la Cuenca del Alto Atoyac y su impacto en la salud
Indice
Artículos del número anterior.
Las suculentas y la vista, una relación inesperada
Noticias de la Academia Mexicana de Ciencias
Desde las redes
Innovaciones tecnológicas en la uam: una mirada desde el doctorado
Tejiendo el futuro: nanotecnología en la industria textil
Las emociones en la toma de decisiones
Una nueva terapia para la leucemia linfoblástica aguda
Linfocitos T: desarrollo, activación y funciones
Paracetamol, un analgésico popular que no es tan inocuo
Entendiendo el tormento de la cefalea en racimos
“Obesos sanos”, un término equivocado
¿Nos predisponen los genes al consumo de drogas?
El rábano: de lo comestible a lo nanotecnológico
Portada
El romero, una planta para el alivio del dolor
Alfred Wallace: científico y militante social
¿Cómo reducir los gases de efecto invernadero de los rumiantes?
La Sierra de Guadalupe, una fuente de biodiversidad en la Cuenca de México
La unión hace la fuerza: historia de los líquenes
Los dientes, una importante fuente de información morfológica y ecológica
Tejer o no tejer... Las diversas estrategias de caza de las arañas
El control de la temperatura corporal: cuestión de vida o muerte
Relojes moleculares: ¿cuándo aparecieron las especies?
Desde el Comité Editorial
Indice
Comunicaciones libres
La ciencia mexicana en las revistas Nature y Science: la última década 
José Antonio del Río y Héctor Daniel Cortés
En el ambiente científico se comenta que la ciencia mexicana ha mostrado un cierto grado de profesionalización y que en estos momentos debemos apuntar los esfuerzos a producir ciencia que trascienda nuestras fronteras y tenga un impacto real en la ciencia internacional. Para tomar cualquier decisión acerca de qué hacer para fomentar este esfuerzo, es necesario tener información concreta sobre el “estado del arte” de la ciencia mexicana. En este sentido apunta
el Atlas de la ciencia mexicana (www.amc.UNAM.mx/atlas. htm) que nos aporta valiosa información con datos específicos. Mientras más información de este tipo esté disponible, las decisiones que tomen la comunidad científica y los políticos tendrán mayores posibilidades de éxito.
Por otro lado, la comunidad científica también reconoce que en el mundo existen revistas que marcan la pauta en el impacto de las investigaciones en el ámbito internacional. En este conjunto, dos de las revistas de mayor reconocimiento en el ambiente científico son Nature y Science. Creemos que la información obtenida de los artículos publicados en esas revistas por científicos laborando en México aportará información relevante para la toma de decisiones de política científica.
En este artículo se presenta el análisis de “minería de citas” (Kostoff y del Río, 2001; del Río y colaboradores, 2002) de los resúmenes de los artículos publicados durante casi una década (de 1995 a principios de 2004) en la revistas Nature y Science en los que algún autor tiene dirección en México o dirección de correspondencia en México. Este periodo no es amplio, pero tampoco es muy corto y sí muy reciente. Seguramente estamos dejando de lado trabajos importantes en algunos campos de la ciencia mexicana; sin embargo, estamos convencidos que si la ciencia mexicana contribuye sustancialmente en alguna área, encontraremos sus hue-
llas en este análisis.
La información se obtuvo de la base de datos del Institute for Scientific Information bajo los siguientes criterios de búsqueda: fuente (Nature AND Science) AND address (Mexico NOT New), esto último para evitar las direcciones en el estado norteamericano de Nuevo México. Con esta búsqueda se obtuvo la información de 110 artículos publicados en estas revistas en casi una década. De este total de artículos, 62 y 48 fueron publicados en Science y Nature, respectivamente. Un punto que debemos hacer notar es que, para un periodo de ocho años, el número de artículos publicados es pobre. El total de artículos publicados en Nature o Science cada año es de 5 mil; de éstos, 2 mil 600 son artículos regulares, cartas de investigación y material editorial. De acuerdo con estudios previos (Kostoff y colaboradores, 2001), con un número de artículos mayor a 50, el conjunto puede ser analizado y del análisis resultante se pueden obtener indicadores del trabajo científico desarrollado en México en el lapso de casi una década. Solamente para comparar, comentemos que los científicos radicados en Argentina, Brasil y España produjeron en esta última década 70, 159 y 391 artículos, respectivamente.
La minería de citas consiste en el conteo de algunos datos de los campos que se obtienen de la base de datos del Institute for Scientific Information bajo la búsqueda indicada arriba; en adición, se usa un conteo de palabras clave para indagar algunos aspectos cualitativos. A continuación se presentan los resultados de este conteo realizado con un programa de computadora expresamente creado para este fin.
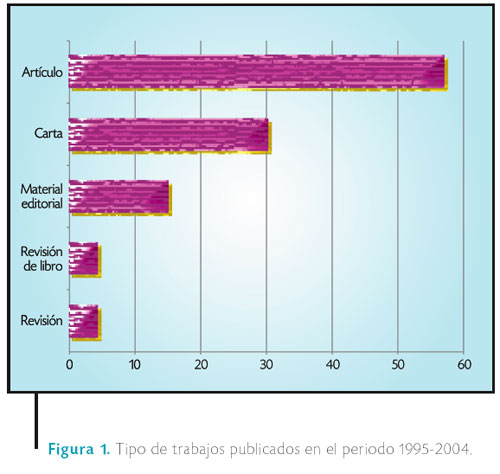
El primer resultado que presentamos es el conteo del tipo de publicación. En estas revistas se aceptan contribuciones como artículos regulares, cartas, material editorial, artículos de revisión y comentarios a libros. En la Figura 1 observamos que del orden del 80 por ciento de los trabajos publicados son artículos regulares o cartas de investigación. Dado el carácter multidisciplinario de estas revistas, no se puede observar la temática, pero en el análisis de minería de textos trataremos de obtener algunos resultados. La presencia de material editorial, artículos de revisión y reseñas de libros también es un punto que merece un análisis más detallado, y puede ser motivo de reflexión.
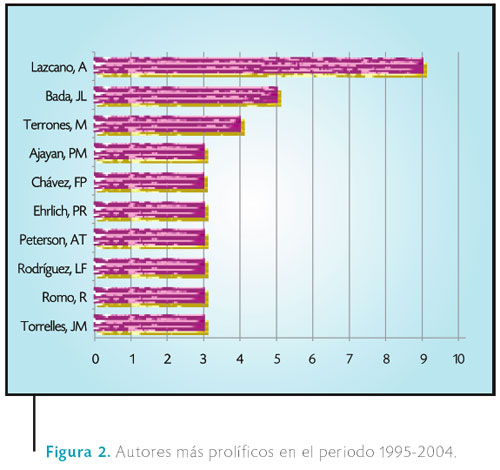
Un segundo resultado que presentamos es el conteo de los autores que más aparecen con trabajos publicados en las revistas Nature y Science. En la Figura 2 se muestran todos los investigadores que aparecen con más de tres artículos y donde un autor tiene una dirección en México en el periodo analizado. Los autores Lazcano (origen de la vida), Rodríguez (astrofísica), y Romo (fisiología celular) laboran en la Universidad Nacional Autónoma de México (UNAM); mientras que Terrones (nanomateriales) labora en el Instituto Potosino de Ciencia y Tecnología; y Chávez en el Monterey Bay Aquarium Research Institute, que colabora con el Centro de Investigación Científica y de Educación Superior de Ensenada (CICESE) y el Centro Interdisciplinario de Ciencias Marinas del Instituto Politécnico Nacional (CICIMAR).
Cabe mencionar que en total se encontraron 642 autores diferentes para los 110 artículos que se analizan. Aquí resalta Lazcano, que ha tenido un promedio de una contribución por año publicada en alguna de estas revistas. Estas contribuciones son cartas, material editorial y revisión de libros. Es importante aclarar que de la lista los apellidos no castellanos son de científicos que colaboran con científicos radicados en México (J. L. Bada con Lazcano; P. M. Ajayan con M. Terrones; los casos de Ehrlich y Peterson son similares al caso de Chávez, ya que colaboran con más de dos científicos mexicanos que no aparecen en esta lista, y Torrelles –español– con Rodríguez), y que su presencia en esta lista indica su grado de colaboración con la ciencia desarrollada en México. Por otro lado, al buscar los nombres en el directorio de miembros de la Academia Mexicana de Ciencias (2004) observamos que el área que cada autor cultiva es diferente. En este análisis no se distingue el orden de aparición de los autores en las publicaciones.
 |
 |
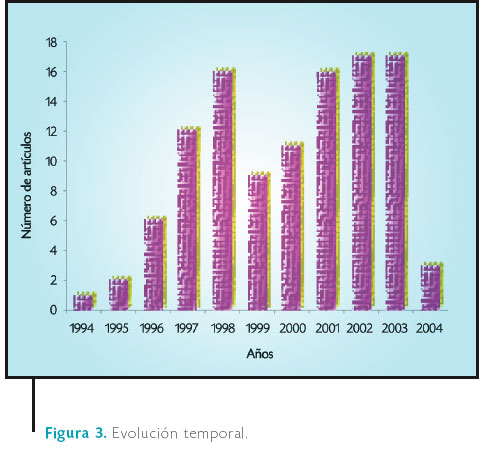
En este tipo de análisis siempre cabe la pregunta acerca de cuál es la tasa por año de publicaciones mexicanas en estas revistas. Del número total de publicaciones en el periodo se podría inferir una tasa del orden de 12 publicaciones anuales en estas revistas por parte de algún científico trabajando en México. En la Figura 3 se muestra la evolución temporal del número de artículos en estos años. Es importante mencionar que aunque la búsqueda se limitó al periodo 1995-2004, se encontró un artículo publicado en 1994, pero reportado al Institute for Scientific Information en 1995. En el año 2004 solamente aparecen tres publicaciones, debido a que la búsqueda fue realizada en el mes de abril de ese año. En la gráfica llama la atención que, a no ser por el valle en los años 1999 y 2000, parece existir una tendencia a estabilizarse en un número aproximado de 17 artículos por año, hecho cuyas causas merecen un análisis más detallado: quizás se deba a la política científica o a la crisis económica. Pero una conducta oscilante en la tasa de producción científica frecuentemente es asociada a un número pequeño de científicos, es decir, a una carencia de masa crítica para mantener una producción científica ya no sobresaliente, sino estable. En otras palabras, las oscilaciones de la producción científica que parecen existir son un indicador de una comunidad no madura en su conjunto que todavía no se consolida. Esto sugeriría que todavía no se alcanza una masa crítica de científicos en México, que permita lograr un mayor impacto en la ciencia internacional. En este punto cabe insistir que la muestra fue tomada en una ventana de tiempo corta, pero muy reciente.
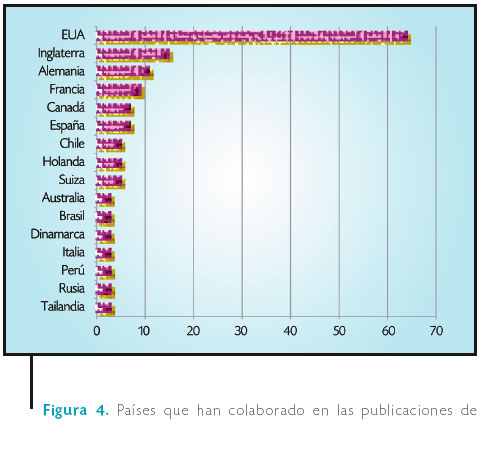
Uno de los aspectos importantes en la publicación de estos trabajos puede ser obtenida del dato de las colaboraciones de los científicos mexicanos con sus colegas en otros países. En la Figura 4 se observan estas colaboraciones cuando se produjeron más de tres trabajos publicados. Se observa una abrumadora colaboración con los Estados Unidos de América. Este resultado muestra la gran dependencia de los trabajos de investigadores en México de la ciencia desarrollada en los Estados Unidos de América. Este país aparece en más del 50 por ciento de los artículos publicados por mexicanos, situación que es más del triple de cualquier otro país. (Para este conteo se toma en cuenta la aparición del país en la dirección de los artículos.) Sorprende la colaboración con Tailandia, que aparece con algún investigador coautor en más de tres artículos.
 |
 |
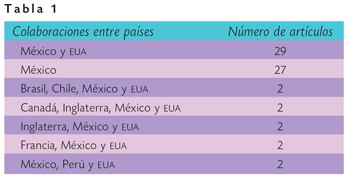
Los aspectos de colaboraciones entre un grupo de países son también importantes. En la Tabla 1 se observan los grupos de países que tienen más de dos artículos en colaboración. Es de hacerse notar que el mayor número de colaboraciones se da entre los Estados Unidos de América y México; de hecho, es mayor el número de colaboraciones con ese país que con cualquier otro incluyendo al propio México. Otro punto es que los artículos publicados solamente por investigadores con dirección en México ocupa el segundo lugar, y representa más del 25 por ciento de las publicaciones en estas revistas. Fuera de esos dos grupos los datos caen dramáticamente a dos o menos artículos en colaboración por grupo de países. En estos 110 artículos participaron investigadores radicados en 41 países diferentes.
 |
 |
 |
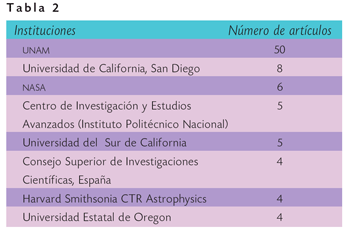
Del análisis bibliométrico también se pueden obtener la relación de las colaboraciones a nivel institución. En la Tabla 2 se presenta el número de artículos donde aparece una institución. Solamente se muestran las instituciones con más de tres artículos publicados en el periodo analizado. En total se encontraron 270 diferentes instituciones participando en estas 110 publicaciones. Dentro de las más prolíficas reconocemos en ellas instituciones de tres países: México, usa y España. En el caso del Consejo Superior de Investigaciones Científicas (CSIC), en España, al buscar la correlación con la Tabla 1 podemos concluir que esta institución colabora con al menos dos grupos de investigación que tienen contacto con México y que publican en Nature o Science.
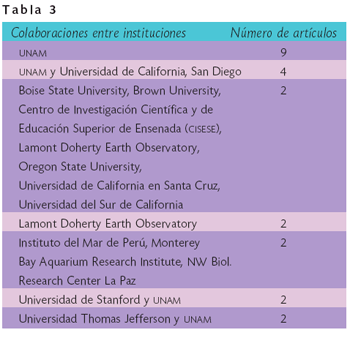
En la Tabla 3 se pueden observar las colaboraciones entre instituciones que produjeron más de un artículo en estas revistas en este periodo de tiempo. En la tabla se muestran las colaboraciones con al menos dos trabajos publicados. Aquí destaca la colaboración entre la Universidad Nacional Autónoma de México y la Universidad de California en San Diego. También se observa que la UNAM es la única institución mexicana que produjo, durante este periodo de tiempo, más de un artículo en estas revistas sin colaboración de otra institución, sea mexicana o extranjera. Esta tabla y la Figura 2 indican que no existe en México un cúmulo de trabajo científico que produzca resultados publicables en estas revistas de forma al menos anual; esta afirmación se sustenta en que la correlación entre las nueve publicaciones de Lazcano y la UNAM no es completa.
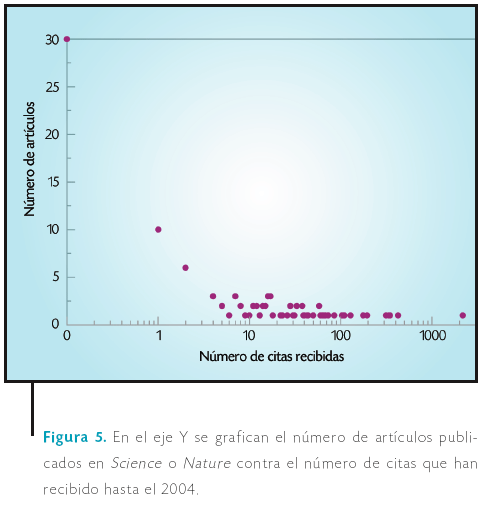
Cuando los científicos publican en revistas como Nature o Science, sus expectativas radican fundamentalmente en la búsqueda de un mayor impacto de su trabajo de investigación. Generalmente esto se contabiliza como el número de citas recibidas por su trabajo publicado. En la Figura 5 se presenta el número de artículos de investigadores en México en relación con el número de citas que han recibido. Se observa que 30 de estos trabajos no han recibido cita alguna, que 10 de ellos sólo han recibido una cita y que 11 han recibido más de 100 citas.
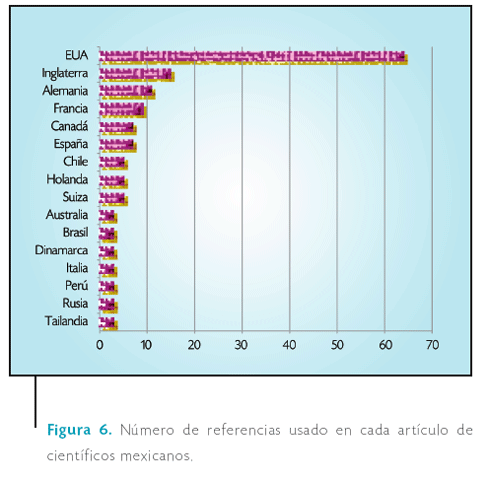
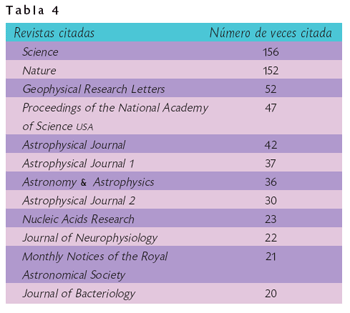
De la base de datos del Institute for Scientific Information se puede obtener otro resultado sobre el número de referencias que utiliza un trabajo realizado por investigadores que laboran en México y que fue publicado en Nature o Science. En la Figura 6 se nota que estos artículos tienen en su mayoría menos de cuarenta referencias, y que más de 30 de ellos usaron menos de 10 referencias para fundamentar su tabajo. De estas referencias, la mayoría de ellas fueron a las propias revistas Nature y Science, como se ve en la Tabla 4. En esta tabla solamente se presentan revistas que recibieron más de 20 citas. De la Tabla 4 se observa que tres revistas de las más referidas son multidisciplinarias, cinco de astronomía y una de geofísica, neurofisiología, genética y bacteriología. De esta tabla se pueden inferir los principales tópicos de las investigaciones analizadas en este trabajo. El total de revistas citadas en los 110 artículos es de 863, lo que manifiesta una gran diversidad de lectura de material científico y reafirma el hecho que hay una dispersión de temas.
En el análisis bibliométrico también se puede obtener la tabla de los artículos más citados; dado que no se encontró ningún artículo citado más de tres veces, esta información no se presenta. Consideramos que un trabajo citado en no más de tres artículos de los analizados no da información útil.
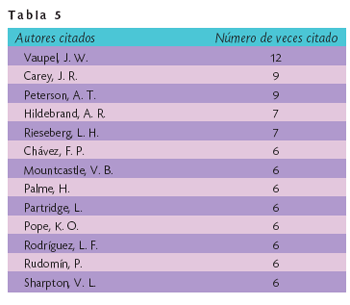
Otro indicador interesante es la tabla de autores más citados en estos trabajos. Estos datos se encuentran en la Tabla 5, donde se presentan solamente los autores que recibieron más de seis citas en el conjunto de 110 trabajos. Por otro lado, también debemos tomar en cuenta que los autores más referidos en los 110 trabajos son coautores de algunos de estos mismos trabajos, pero no realizan su trabajo de investigación en México; asimismo, notamos que el número de coincidencias entre autores del trabajo y autores citados es pobre (de la lista solamente hay dos nombres de investigadores que laboran en México: Rodríguez, en la UNAM, y Rudomín, que labora en el Centro de Investigación y Estudios Avanzados).
 |
 |
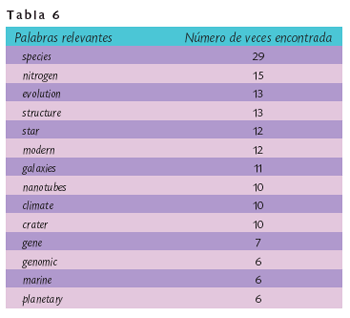 |
La Tabla 5 indica que la diversidad de temas es grande. En este análisis solamente se considera al primer autor del trabajo referido. En este punto debemos hacer notar que algunos científicos mexicanos no son citados de forma estándar y por lo tanto se pueden perder sus citas en búsquedas generales y automatizadas.
A continuación presentamos los resultados de un sencillo análisis de texto. Para ello se analizaron los resúmenes que provee la base de datos. En solamente hay resúmenes en los artículos regulares. Por lo tanto, fueron analizados bajo este esquema solamente 59 trabajos.
Usamos una técnica para definir las palabras clave de un texto (Ortuño y colaboradores, 2002). Este método se basa en medir la distancia entre la aparición de las palabras en un texto y el cálculo de su desviación estándar. Una desviación estándar normalizada mayor a uno indica que la distribución de esa palabra en el texto es no aleatoria. De esta manera se puede determinar que las palabras que cumplan con esta característica son relevantes en el texto analizado. Esta idea se sustenta en que la desviación estándar es un indicador análogo a la entropía, y en algunas ocasiones puede jugar su papel (Reiss y colaboradores, 1986) como medida del orden (o, inversamente, del desorden).
En el análisis de una sola palabra encontramos algunas palabras relevantes que aparecen en la Tabla 6. Las palabras también corroboran la temática que indicaban las revistas más citadas. Sin embargo, la observación más importante es que hay una gran dispersión en los 110 trabajos, lo cual confirma que no existe un campo de liderazgo internacional en el quehacer científico mexicano.
Podemos inferir que la ciencia en México todavía no llega a tener una masa crítica de investigadores que produzca conocimiento constante
Conclusiones
Con este análisis se observa que existen temáticas que han logrado publicar trabajos en las revistas Science y Nature. Sin embargo en este análisis no aparece una línea de investigación con una clara producción sistemática que indique su relevancia en el contexto internacional. De lo anterior podemos inferir que la ciencia en México todavía no llega a tener una masa crítica de investigadores que produzca conocimiento constante.
Claramente el análisis aquí presentado tiene limitaciones y no rescata todos los trabajos relevantes de la ciencia mexicana. Como ya se ha mencionado, entre otras posibles limitaciones del trabajo se encuentra que la ventana de análisis es limitada. Esto último implica la necesidad de realizar más estudios similares a éste por área de especialidad, analizando revistas de alto impacto (por ejemplo: Cell, Physical review, Trends in ecology and evolution) y apoyar con mayor decisión trabajos como el Atlas de la ciencia en México, que en conjunto nos darán una visión de la situación real de la ciencia en México y aportará elementos para la toma de decisiones.
Por otro lado, dada la situación que refleja este análisis consideramos que todavía la población de científicos mexicanos no ha alcanzado un número suficiente para generar conocimiento relevante en forma sostenida, y proponemos que se dediquen mayores esfuerzos a crear ese número crítico de científicos profesionales para que en el camino se puedan ir gestando áreas de investigación mexicana que puedan tener incidencia en el contexto internacional.
Agradecimientos
Este trabajo fue parcialmente apoyado por el Consejo Nacional de Ciencia y Tecnología y el Gobierno del Estado de Morelos, bajo el proyecto FOMIX 9250. También hacemos patente nuestro agradecimiento a Luis Felipe Rodríguez por habernos sugerido analizar las publicaciones de mexicanos en Nature y Science.
Bibliografía
Academia Mexicana de Ciencias (2004), Directorio, CD-ROM.
Academia Mexicana de Ciencias, Atlas de la ciencia en México, www.amc.unam.mx/atlas.htm
Kostoff, R. N. y J. A. del Río (2001), “The impact of physics research”, Physics World, pág. 47-51.
Kostoff, R. N., J. A. del Río, J. A. Humenik, E. O. García y A. M. Ramírez (2001), “Citation mining: integrating text mining and bibliometrics for research user profiling”, J. Am. Soc. Inf. Sci. Techn. 52, 1148.
Ortuno, M., P. Carpena, P. Bernaola-Galván, E. Muñoz, A. M. Somoza (2002), “Keyword detection in natural languages and DNA, Europhysics Letters 57, 759.
Reiss, H., A. D. Hammerich y E. W. Montroll (1986), “Thermodynamics of non-physical systems”, J. Stat. Phys. 42, 647.
Del Río, J. A, R. N. Kostoff, E. O. García, A. M. Ramírez y J. A. Humenik (2002), “Phenomenological approach to profile impact of scientific research: citation mining”, Adv. Complex Syst. 5, 19. También disponible en: http://arXiv.org/abs/physics/0112047.
Jesús Antonio del Río Portilla es investigador titular del Centro de Investigación en Energía de la UNAM en Temixco, Morelos. Doctor en Ciencias por la UNAM, es miembro de las Academias Mexicana de Ciencias, de Ingeniería y de Ciencias de Morelos. Sus líneas de investigación versan sobre termodinámica de procesos irreversibles, nanoestructuras y sistemas complejos.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
Héctor Daniel Cortés González es ingeniero en sistemas computacionales del Instituto Tecnológico de Mérida. En 2004 recibió la certificación de Ingeniero RedHat (rhce). Actualmente trabaja en la Unidad de Cómputo del Centro de Investigación en Energía de la Universidad Nacional Autónoma de México (UNAM) y colabora en proyectos de minería de citas y desarrollo de software libre.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
Aplicaciones exitosas
del diseño de fármacos utilizando
métodos computacionales 
José Luis Medina Franco
Los fármacos actúan produciendo cambios en algún proceso o función fisiológica. Muchos ejercen su efecto al interactuar específicamente con alguna estructura macromolecular del organismo. Para referirse a esta macromolécula, Paul Erlich propuso el término “receptor” a principios del siglo pasado. De esta forma, la interacción de cada fármaco con su respectivo receptor o sitio de acción inicia los cambios bioquímicos y fisiológicos que son característicos de ese fármaco.
Un ejemplo de un sitio de acción son los de las enzimas, proteínas especializadas en acelerar reacciones bioquímicas. La interacción de un fármaco con su blanco molecular se ha comparado en forma muy sencilla con lo que sucede entre una llave y una cerradura. En este caso, la llave encaja en la cerradura para producir una acción: abrir o cerrar esa cerradura.
Cuando se realiza la investigación de nuevos fármacos, nuevos o modificados a partir de moléculas existentes, primero se recopila toda la información posible sobre los procesos biológicos asociados a la enfermedad bajo estudio. En diversas ocasiones se logra identificar a un blanco molecular específico sobre el cual puede actuar un fármaco. Posteriormente se trata de determinar la estructura tridimensional de ese blanco para conocer con detalle el sitio de unión sobre el cual se pretende que interactúe el fármaco. Volviendo a la analogía de la llave con la cerradura, si se quiere fabricar una llave que entre en una cerradura determinada, esto será más sencillo si se conoce la forma de la cerradura.
 Una de las técnicas más empleadas actualmente para conocer la estructura tridimensional de macromoléculas es la cristalografía de rayos X. Otra técnica experimental es la resonancia magnética nuclear. A pesar de que el conocimiento de la estructura tridimensional es muy valioso para entender los mecanismos de acción de fármacos y diseñar a nuevas moléculas, en muchas ocasiones ésta se desconoce. Esto se debe, en parte, a la dificultad que presenta el uso de este tipo de técnicas. En estos casos, se puede recurrir a métodos computacionales, por ejemplo, a la metodología conocida como “modelado por homología”.
Una de las técnicas más empleadas actualmente para conocer la estructura tridimensional de macromoléculas es la cristalografía de rayos X. Otra técnica experimental es la resonancia magnética nuclear. A pesar de que el conocimiento de la estructura tridimensional es muy valioso para entender los mecanismos de acción de fármacos y diseñar a nuevas moléculas, en muchas ocasiones ésta se desconoce. Esto se debe, en parte, a la dificultad que presenta el uso de este tipo de técnicas. En estos casos, se puede recurrir a métodos computacionales, por ejemplo, a la metodología conocida como “modelado por homología”.
Los grupos de investigación alrededor del mundo que determinan la estructura tridimensional de macromoléculas almacenan sus resultados en la base de datos pública llamada Protein Data Bank (Banco de Datos de Proteínas, ver “páginas en internet de interés” al final del artículo). Actualmente, esta base de datos contiene información de aproximadamente cuarenta y un mil estructuras, y se actualiza constantemente. Dentro de esta base de datos cada estructura tridimensional tiene asociado un código de cuatro caracteres (llamado código PDB) que la identifica en forma única. Para la visualización en tercera dimensión de estas macromoléculas hay diversas herramientas en internet a las que se puede acceder desde la misma página del Protein Data Bank. También hay diversos programas computacionales gratuitos (ver “páginas en internet de interés”). Algunos de estos programas permiten incluso realizar cierto manejo de las estructuras y cálculos teóricos.
Al uso de la información estructural del blanco molecular para diseñar fármacos se le llama “diseño de fármacos basado en la estructura del receptor”. En este tipo de diseño las computadoras son una herramienta muy valiosa; casi imprescindible. Los programas computacionales permiten no solamente visualizar las estructuras de macromoléculas sino también hacer cálculos teóricos y predicciones de la afinidad que tendrá una molécula, aun hipotética, con el sitio de unión. Esta capacidad de predicción ayuda a proponer hipótesis y planear experimentos para hacerlos más enfocados y sistemáticos. La importancia y gran uso de las computadoras en el diseño de fármacos ha dado origen al área de investigación conocida como “diseño de fármacos asistido por computadora”.
Diseño de fármacos basado en la
estructura del receptor
Se define como la búsqueda de moléculas que encajen en el sitio de unión de un blanco molecular de manera que puedan formar interacciones favorables. Uno de los aspectos más importantes del diseño basado en la estructura del receptor busca mejorar las interacciones que ocurren entre una molécula y su blanco molecular. Esto es, se parte de un fármaco conocido pero que no tiene el efecto requerido; sin embargo, la estructura tridimensional del complejo molécula-blanco proporciona información para realizar la optimización. Con este propósito se recurre a uno de los métodos computacionales más usados en este campo: el “acoplamiento molecular automatizado” (Kitchen y colaboradores, 2004). Este método consiste en calcular con la computadora cuál es la posición más favorable que tendría una molécula con el blanco molecular. A partir del resultado se propocho, se puede trabajar con moléculas hipotéticas que no se tienen en el laboratorio, o que aún no han sido preparadas. En la industria farmacéutica es común trabajar con las llamadas “bibliotecas virtuales”: colecciones de miles o millones de moléculas hipotéticas. Sin embargo, después de hacer los cálculos, estas moléculas virtuales pueden ser fabricadas y evaluadas biológicamente.
 Otro aspecto del diseño basado en la estructura del receptor es diseñar, desde cero, una molécula que tendrá interacciones favorables con el blanco molecular. A este proceso se le conoce como “diseño de novo” y ha tenido avances notables en el diseño de fármacos (Schneider y Fechner, 2005). En general, esta estrategia consiste en construir, fragmento a fragmento, una molécula que según los cálculos tendrá interacciones favorables con el receptor. Los probables sitios de unión pueden conocerse a través de experimentos (por ejemplo, mutaciones dirigidas o resonancia magnética nuclear) o con la ayuda de métodos computacionales.
Otro aspecto del diseño basado en la estructura del receptor es diseñar, desde cero, una molécula que tendrá interacciones favorables con el blanco molecular. A este proceso se le conoce como “diseño de novo” y ha tenido avances notables en el diseño de fármacos (Schneider y Fechner, 2005). En general, esta estrategia consiste en construir, fragmento a fragmento, una molécula que según los cálculos tendrá interacciones favorables con el receptor. Los probables sitios de unión pueden conocerse a través de experimentos (por ejemplo, mutaciones dirigidas o resonancia magnética nuclear) o con la ayuda de métodos computacionales.
Aplicaciones exitosas del diseño
de fármacos asistido por computadora
Para muchas enfermedades se conocen estructuras tridimensionales de potenciales sitios de acción de fármacos. En diversas ocasiones los cálculos computacionales han tenido un papel muy importante en la investigación de moléculas que se unen a estos blancos y que actualmente se encuentran en uso clínico. Por ejemplo, el diseño de fármacos asistido por computadora ya ha tenido contribuciones notables en el tratamiento del síndrome de la inmunodeficiencia adquirida, en infecciones por el virus de la influenza y en el tratamiento del glaucoma.
Captopril: primer diseño basado en el receptor
La llamada enzima convertidora de angiotensina participa en dos procesos que parecen ser importantes en la regulación presión arterial. Por una parte, acelera la conversión de la angiotensina I en angiotensina II, que es un vasoconstrictor potente. Por otra parte inactiva a la bradiquinina, que es un vasodilatador. Por tanto, la enzima convertidora de angiotensina es un blanco molecular adecuado para el tratamiento de pacientes con hipertensión.
La investigación de inhibidores de la enzima convertidora de angiotensina surgió a partir de una sustancia natural obtenida de una víbora venenosa del Brasil. Al momento de iniciarse los estudios, en la década de los setenta, no se conocía la estructura tridimensional de esta enzima. Sin embargo, sí se conocía la estructura de una enzima parecida, la carboxipeptidasa A de bovino. Utilizando a esta estructura como modelo y siguiendo una metodología que actual-mente se denomina “diseño de análogo activo”, investigadores de la compañía Squibb desarrollaron el captopril, aprobado en Estados Unidos para su uso clínico en 1981. Aunque el captopril no se desarrolló utilizando cálculos computacionales se puede considerar como el primer ejemplo de un fármaco diseñado basado en la estructura del receptor. Recientemente se dio a conocer la estructura del captopril unido a la enzima convertidora de angiotensina (Figura 1), confirmando la hipótesis bajo la cual fue diseñado.
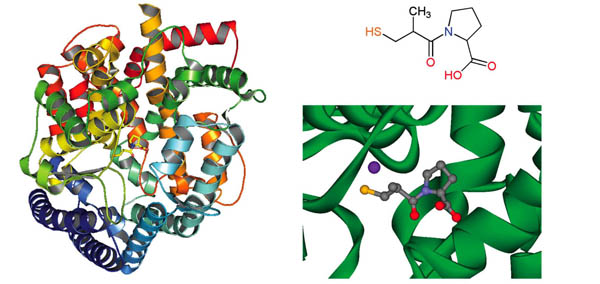
Figura 1. Estructura tridimensional de la enzima convertidora de angiotensina (izquierda) (código PDB: 1UZF), estructura química del captopril (derecha arriba) y detalle de la unión con la enzima.
Fármacos para el tratamiento del glaucoma
La anhidrasa carbónica II humana es una enzima que facilita la hidratación del dióxido de carbono para formar bicarbonato. Aunque diversas anhidrasas carbónicas se localizan en varios órganos, tejidos y células del cuerpo, la anhidrasa carbónica II es de especial interés porque su actividad está asociada a un incremento en la presión intraocular, produciendo de esta manera el glaucoma. Por lo tanto, los inhibidores de la anhidrasa carbónica son atractivos para el tratamiento de esta enfermedad.
Desde hace muchos años se ha utilizado a la metazolamida para el tratamiento del glaucoma. Sin embargo, debido a que este fármaco se administra en forma oral, causa efectos secundarios al inhibir la anhidrasa carbónica que se encuentra en otras partes del cuerpo. Por esta razón, era deseable contar un fármaco que pudiese administrarse en forma tópica (local y externa).
A mediados de la década de los ochenta la compañía Merck obtuvo por cristalografía de rayos X la estructura de la anhidrasa carbónica unida a una molécula que denominaron MK-927. A partir de esta estructura, de cálculos de la energía de diversas moléculas similares a la MK-927 y de estudios cristalográficos, se diseñó a la dorzolamida (Figura 2), un inhibidor potente de la anhidrasa carbónica. Su uso clínico se aprobó en 1994, con el nombre de Trusopt® y fue el primer inhibidor de la anhidrasa carbónica que se logró formular como solución oftálmica y, por tanto, administrarse en forma tópica. Cuatro años después se aprobó a la brinzolamida (Azopt®) que tiene una estructura química muy parecida a la dorzolamida y también se administra en forma tópica (Figura 2).
Fármacos para el tratamiento
del glaucoma
Otro ejemplo del éxito del diseño de fármacos utilizando la estructura del receptor y métodos computacionales es el desarrollo del relenza, fármaco empleado para el tratamiento de la infección causada por el virus de la influenza. Utilizando la estructura tridimensional de la enzima sialidasa (llamada también neuraminidasa, un blanco molecular para atacar al virus), y métodos computacionales, se propuso la estructura de inhibidores potentes de esta enzima (von Itzstein y colaboradores, 1993). La estrategia computacional consistió principalmente en el análisis gráfico de la estructura de la sialidasa y el uso del programa GRID. Este programa, muy utilizado en el diseño de novo de fármacos, es especialmente valioso para encontrar posibles sitios de unión en una macromolécula. Uno de los compuestos diseñados en la compañía hoy llamada GlaxoSmithKline, fue el relenza que se aprobó para su uso clínico en 1999 con el nombre de Zanamivir® (Figura 3). Otro fármaco para el tratamiento de la influenza es el oseltamivir (Tamiflu®), también aprobado en 1999 y hoy comercializado por la compañía Roche. La investigación de este fármaco, que está relacionado con el relenza, también se hizo apoyándose en la estructura tridimensional de la sialidasa.
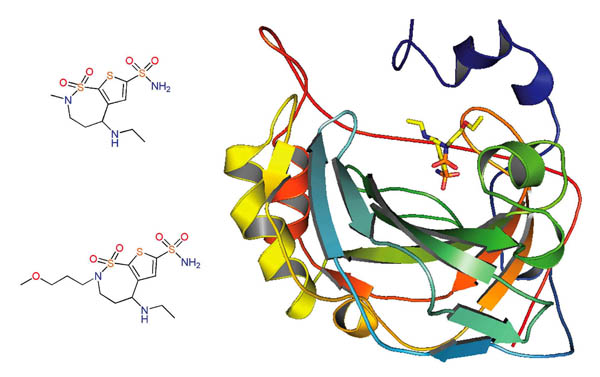
Figura 2. Estructura química de la dorzolamida (Trusopt ) (izquierda mensional de la anhidrasa carbónica (código PDB: 1A42).
Fármacos contra el SIDA
El mayor número de aplicaciones exitosas del diseño basado en la estructura del receptor con la ayuda de métodos computacionales ha ocurrido hasta ahora en el campo del tratamiento del síndrome de la inmunodeficiencia adquirida (sida). Poco después de que se detectaran los primeros casos a principios de la década de los ochenta, se encontró que el virus de la inmunodeficiencia humana (VIH) es el causante de esta enfermedad.
Hay diversos blancos moleculares sobre los cuales pueden interactuar fármacos para detener la infección causada por este virus. Uno de ellos es la enzima proteasa del VIH, que interviene en la maduración de las partículas virales. La estructura tridimensional de esta enzima se dio a conocer a finales de la década de los ochenta. Hacia 1990 se reportó una de las primeras aplicaciones del diseño basado en la estructura de esta enzima con el desarrollo del compuesto entonces llamado Ro 31-8959. Este diseño culminó cinco años después con la aprobación del saquinavir como el primer inhibidor de la proteasa del VIH utilizado en el tratamiento del sida (Figura 4). A partir de la estructura tridimensional de esta enzima se han diseñado y aprobado para su uso clínico ocho inhibidores de la proteasa de VIH. El inhibidor de más reciente aprobación es el tipranavir (Aptivus®), cuyo uso clínico se autorizó en Estados Unidos el 22 de junio de 2005 (Figuras 4 y 5). El uso de métodos computacionales, aunado al análisis estructural y síntesis química, ha participado en forma muy importante en la investigación que produjo estos fármacos. Los estudios computacionales han involucrado, generalmente, análisis gráficos de las estructuras tridimensionales y cálculos de energía. Resulta muy interesante el desarrollo del indinavir (Figura 4, referido originalmente por la compañía Merck con el código L-735,524), que involucró la predicción correcta de la actividad biológica de diversas moléculas utilizando cálculos teóricos (Holloway y colaboradores, 1995).
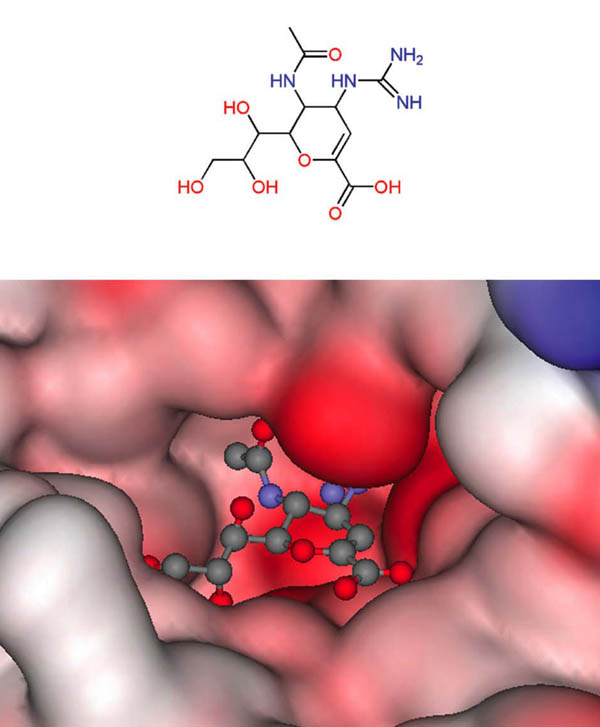
Figura 3. Estructura química del relenza (Zanamavir®) y detalle del sitio de unión con la enzima sialidasa del virus de la influenza (código PDB: 1A4G).
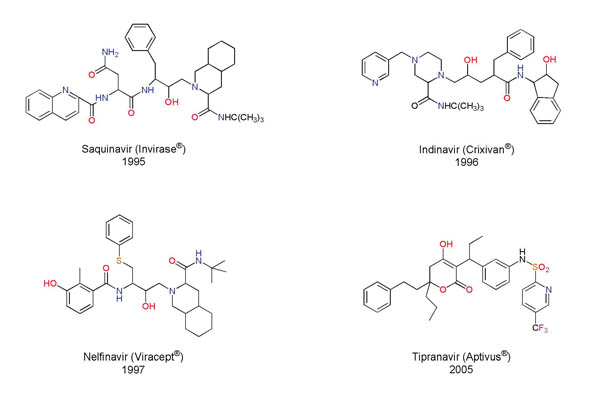
Figura 4. Ejemplos de fármacos empleados en el tratamiento del sida que inhiben a la proteasa del VIH. Se muestra el nombre comercial entre paréntesis y el año de aprobación.
Otro de los blancos moleculares de gran interés para atacar al virus del sida es la enzima transcriptasa inversa. Esta enzima, que convierte el ácido ribonucleico (ARN) que forma los genes del VIH en ácido desoxirribonucleico (ADN), participa en la replicación de las partículas virales dentro de la célula huésped. En México se está trabajando, mediante técnicas computacionales, en el diseño basado en la estructura del receptor de inhibidores de esta enzima. Utilizando acoplamiento molecular automatizado se han estudiado las interacciones que puede haber entre diversas moléculas y la transcriptasa inversa del VIH (Medina-Franco y colaboradores, 2004a). Partiendo de estos cálculos y otros estudios computacionales se han diseñado moléculas que son inhibidores prometedores de este blanco molecular (Medina-Franco y colaboradores, 2004b).
El futuro del diseño de fármacos mediante técnicas computacionales
Además de los fármacos diseñados con la ayuda de métodos computacionales se ha determinado la estructura tridimensional de diversos fármacos en uso clínico, obtenidos o diseñados por otros métodos, y de sus respectivos blancos moleculares. Algunos ejemplos son la aspirina unida a la enzima cycloooxigenasa I (aunque se sabe que también se une a la cycloooxigenasa II; de ahí sus efectos secundarios de irritación del estómago, gastritis y daño renal), el imatinib (Gleevec®) empleado en el tratamiento de ciertos tipos de enfermedades malignas (específicamente la leucemia mieloide crónica y los tumores del estroma gastrointestinal) y la atorvastatina (Lipitor®), usada en el tratamiento de la hiperlipidemia. Esta información está siendo de gran utilidad para el diseño de nuevos fármacos.
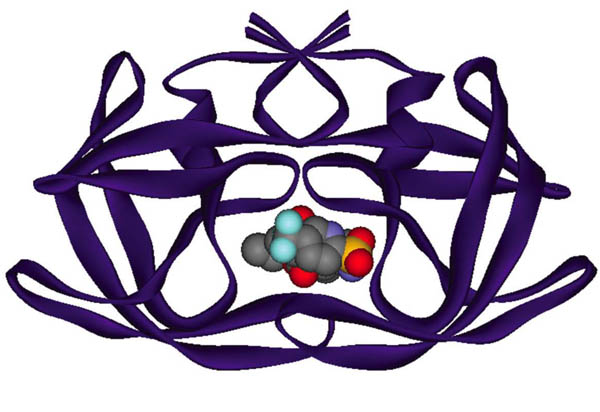
Figura 5. Estructura de la proteasa del VIH unida al tipranavir (Aptivus®) (código PDB: 1D4S).
Hoy el uso de la información estructural de los blancos moleculares y métodos computacionales es una práctica común en la industria farmacéutica y en instituciones académicas y de gobierno de todo el mundo. Esto ha sido favorecido por el fácil acceso a muchos programas que son potentes y tienen un costo muy bajo o incluso son gratuitos. Asimismo, la capacidad de los equipos de cómputo va en un aumento vertiginoso, y los costos de estos equipos son cada vez más accesibles. Como consecuencia, además de los ejemplos mostrados de diseño exitoso de fármacos con métodos computacionales, hay muchos casos en que estas metodologías están aportando información muy importante a la investigación. En México, además de los estudios mencionados con la transcriptasa inversa, se ha realizado acoplamiento molecular automatizado con la isomerasa de triosasfosfato (Espinoza-Fonseca y Trujillo-Ferrara, 2004) y con la 3-hidroxi-3-metilglutaril-coenzima A reductasa (Medina-Franco y colaboradores, 2005) para contribuir en el diseño de compuestos antiparasitarios para combatir el colesterol en la sangre (hipocolesterolemiantes), respectivamente. Otras aplicaciones se extienden a diversas enfermedades incluyendo el cáncer, el sida, la enfermedad de Alzheimer, la hipertensión y la artritis, entre muchas otras. Estas aplicaciones, así como los avances en el desarrollo de métodos y programas computacionales para el diseño de fármacos, pueden encontrarse en artículos publicados en revistas como Journal of Medicinal Chemistry, Journal of Computer-Aided Molecular Design, Journal of Chemical Information and Modeling, Bioorganic and Medicinal Chemistry, Current Computer-Aided Drug Design, Nature Reviews Drug Discovery y Science, entre otras. Sin duda, en un futuro cercano veremos más fármacos desarrollados con la ayuda de métodos computacionales.
Conclusión
Las computadoras se han convertido en una poderosa herramienta para generar modelos de fármacos interactuando con biomoléculas. Estos modelos ayudan a entender el mecanismo de acción de los fármacos, proponer modificaciones para mejorar su efecto terapéutico y diseñar nuevas moléculas. Hoy en día, ya hay diversos fármacos en uso clínico que fueron diseñados con la ayuda de métodos computacionales, muchos de ellos para el tratamiento del sida.
Agradecimientos
Agradezco a la doctora Karina Martínez (Universidad de Arizona) y al doctor Heriberto Medina (Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán) sus valiosos comentarios. Dedico este trabajo a la Fundación Lorena Alejandra Gallardo en sus veinticinco años de trabajo incansable en el apoyo a estudiantes mexicanos.
 Para saber más
Para saber más
Espinoza-Fonseca, L. M. y J. G. Trujillo-Ferrara (2004), “Exploring the possible binding sites at the interface of triosephosphate isomerase dimer as a potential target for anti-tripanosomal drug design”, Bioorganic and Medicinal Chemistry Letters, 14, 3151-3154.
Gohlke, H. y G. Klebe (2002), “Approaches to the description and prediction of the binding affinity of small-molecule ligands to macromolecular receptors”, Angewandte Chemie International Edition, 41, 2644-2676.
Holloway, M. K. y colaboradores (1995), “A priori prediction of activity for HIV-1 protease inhibitors employing energy minimization in the active site”, Journal of Medicinal Chemistry, 38, 305-317.
Jorgensen, W. L. (2004), “The many roles of computation in drug discovery”, Science, 303, 1813-1818.
Kuntz, I. D. (1992), “Structure-based strategies for drug design and discovery”, Science, 257, 1078-1082.
Kitchen, D. B. y colaboradores (2004), “Docking and scoring in virtual screening for drug discovery: methods and applications”, Nature Reviews Drug Discovery, 3, 935-949.
Medina-Franco, J. L. y colaboradores (2004a), “Docking-based CoMFA and CoMSIA studies of non-nucleoside reverse transcriptase inhibitors of the pyridinone derivative type”, Journal of Computer-Aided Molecular Design, 18, 345-360.
Medina-Franco, J. L. y colaboradores (2004b), “Flexible docking of pyridinone derivatives into the non-nucleoside inhibitor binding site of HIV-1 reverse transcriptase”, Bioorganic and Medicinal Chemistry, 12, 6085-6095.
Medina-Franco, J. L. y colaboradores (2005), “Molecular docking of the highly hypolipidemic agent ?-asarone with the catalytic portion of HMG-CoA reductase”, Bioorganic and Medicinal Chemistry Letters, 15, 989-994.
Schneider, G. y U. Fechner (2005), “Computer-based de novo design of drug-like molecules”, Nature Reviews Drug Discovery, 4, 649-663.
von Itzstein, M. y colaboradores (1993), “Rational design of potent sialidase-based inhibitors of influenza virus replication”, Nature, 363, 418-423.
Páginas en internet de interés
Protein Data Bank, banco de datos de blancos moleculares y otras estructuras tridimensionales: http://pdbbeta.rcsb.org
Algunos programas gratuitos para visualizar estructuras moleculares en tercera dimensión:Chimera:
http://www.cgl.ucsf.edu/chimera/;
Molscript:
http://www.avatar.se/molscript/;
Pymol:
http://pymol.sourceforge.net/;
Raster 3D:
http://www.bmsc.washington.edu/raster3d/. (En la página del Protein Data Bank puede accederse directamente a los visualizadores King Viewer, Jmol Viewer, WebMol Viewer, entre otros.)
Programa de cómputo gratuito para dibujar estructuras químicas: ACD Labs Chemsketch: http://www.acdlabs.com/download/
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.






